Chưa có sản phẩm trong giỏ hàng.
Tin tức, Công nghệ & sản phẩm mới, Khoa học và đời sống
Sáng tỏ cấu trúc bậc bốn của PieE, một loại enzyme phức tạp liên quan đến tổng hợp kháng sinh
Thí nghiệm SEC-SAXS trên thiết bị BioXolver hãng Xenocs trong phòng thí nghiệm kết hợp với một số kỹ thuật khác làm sáng tỏ cấu trúc bậc bốn của PieE, một loại enzyme phức tạp liên quan đến tổng hợp kháng sinh
Các nhà nghiên cứu từ Đại học Laval đã thành công thiết lập thành công thí nghiệm SEC-SAXS trên thiết bị BioXolver hãng Xenocs trong phòng thí nghiệm tại Structural Biology Platform của Đại học Montréal – Canada để làm sáng tỏ cấu trúc của PieE. Enzyme này tham gia vào quá trình tổng hợp kháng sinh piericidin. Nó tạo điều kiện thuận lợi cho quá trình hydroxyl hóa chất nền với sự trợ giúp của NADPH, O₂, PAD và sự hình thành chất trung gian C4a-hydroperosxyflavin. Điều thú vị là, enzyme này sở hữu một hệ thống kiểm soát để ngăn chặn quá trình oxy hóa NADPH và giảm FAD trong điều kiện không có cơ chất. Các công trình xuất bản hiện tại M.S. Manenda et al.[1] đã làm sáng tỏ cơ sở phân tử của hệ thống điều khiển này với sự trợ giúp của Sắc ký loại trừ kích thước kết hợp (SEC – Size-Exclusion Chromatography) với tán xạ ánh sáng đa góc (SEC-MALS) và tán xạ tia X góc nhỏ (SAXS) kết hợp với tinh thể học protein và xét nghiệm hoạt động của enzyme.
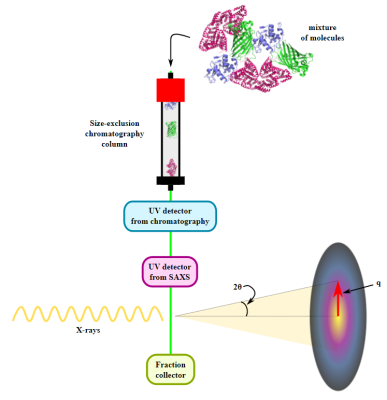
Sử dụng phương pháp tinh thể học protein, PieE đã được chứng minh là tạo ra cấu trúc hexameric ở ba dạng tinh thể khác nhau. Sau đó, SEC-MALS và SEC-SAXS được sử dụng để xác định khối lượng phân tử tuyệt đối và xác nhận sự hiện diện của trạng thái oligomeric trong dung dịch. Hơn nữa, SAXS cung cấp thêm thông tin về cấu trúc: bán kính hồi chuyển (Rg) và kích thước tối đa (Dmax) của enzyme trong dung dịch được xác định bằng kỹ thuật này. Hình dạng 3D của enzyme được tái tạo bằng mô phỏng ab initio và các nhà nghiên cứu đã tạo ra một mô hình nguyên tử giả lập (hình 1) cho thấy sự phù hợp tốt về cả hình dạng và thể tích với cấu trúc tinh thể.

Sự kết hợp của các kỹ thuật này đã cung cấp một cách xác định chắc chắn về cấu trúc của PieE trong dung dịch và ở dạng tinh thể, tạo cơ sở vững chắc cho sự hiểu biết sau này về cơ chế xúc tác. Một sự kết hợp giữa tinh thể học ở các trạng thái khác nhau và các thử nghiệm hoạt động của enzyme ở trạng thái nguyên bản và đột biến của PieE; sau đó đã được sử dụng để khám phá các bước quan trọng và cấu trúc thiết yếu trong chu trình xúc tác có tầm quan trọng đối với toàn bộ nhóm monooxygenase phụ thuộc flavin của Nhóm A, tiết lộ thêm một hình dạng (sliding conformation) tồn tại giữa các hình dạng cổ điển IN và OUT cơ bản.
Chi tiết về cách thiết lập thí nghiệm SEC-SAXS, chuẩn bị mẫu và phân tích dữ liệu trên thiết bị BioXolver hãng Xenocs trong phòng thí nghiệm tại Structural Biology Platform của Đại học Montréal – Canada, xem tại đây!
Nguyễn Tiến Dũng tổng hợp và dịch.
Nguồn: Xenocs.com
Đọc chi tiết công trình của nhóm tác giả trên tại đây:
[1] This research was originally published in the Journal of Biological Chemistry. Manenda MS, Picard M-È, Zhang L, et al. Structural analyses of the group A flavin-dependent monooxygenase PieE reveal a sliding FAD cofactor conformation bridging OUT and IN conformations. J Biol Chem. 2020; 295, 4709-4722 © the American Society for Biochemistry and Molecular Biology.
Và
Size-exclusion chromatography small-angle X-ray scattering of water soluble proteins on a laboratory instrument. Bucciarelli, et al. J. Appl. Cryst. (2018). 51, 1623-1632, https://doi.org/10.1107/S1600576718014462
- Sách trắng – Ứng dụng kỹ thuật Tán xạ tia X góc nhỏ (SAXS) để nghiên cứu, phát triển và bào chế thuốc
- Tìm hiểu thêm thiết bị tán xạ tia X được sử dụng cho ứng dụng trên tại đây!
- Cập nhật những tin tức KHCN & Đời sống mới nổi bật trên thế giới sử dụng kỹ thuật tán xạ tia X góc nhỏ
Một số tin tức KHCN liên quan:
- Lựa chọn, phân tích đặc tính sinh-lý và cấu trúc kháng thể nano tổng hợp giúp vô hiệu hoá hiệu quả vi rút Sars-coV-2
- Tia X có thể giúp test nhanh COVID-19 tin cậy và nhanh chóng hơn trở thành hiện thực như thế nào
- Thiết bị tia X mới tại KU Food kiểm tra bít tết thực vật trong quá trình chiên
- Tìm hiểu thực phẩm ở cấp độ phân tử bằng tia X để tạo ra Snack ngon hơn bao giờ hết trong tương lai
- Tia X sẽ tạo ra chế độ ăn dựa trên thực vật trong tương lai ngon hơn

